Selenate (PAMDB004318)
| Record Information | |||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Version | 1.0 | ||||||||||||||||||||||||||||||||||||||||||||||||
| Update Date | 1/22/2018 11:54:54 AM | ||||||||||||||||||||||||||||||||||||||||||||||||
| Metabolite ID | PAMDB004318 | ||||||||||||||||||||||||||||||||||||||||||||||||
| Identification | |||||||||||||||||||||||||||||||||||||||||||||||||
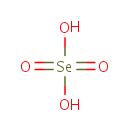
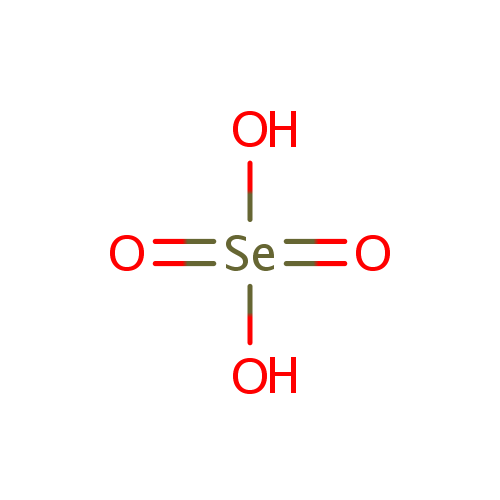
| Name: | Selenate | ||||||||||||||||||||||||||||||||||||||||||||||||
| Description: | A divalent inorganic anion obtained by removal of both protons from selenic acid | ||||||||||||||||||||||||||||||||||||||||||||||||
| Structure | |||||||||||||||||||||||||||||||||||||||||||||||||
| Synonyms: |
| ||||||||||||||||||||||||||||||||||||||||||||||||
| Chemical Formula: | H2O4Se | ||||||||||||||||||||||||||||||||||||||||||||||||
| Average Molecular Weight: | 144.97 | ||||||||||||||||||||||||||||||||||||||||||||||||
| Monoisotopic Molecular Weight: | 145.91183038 | ||||||||||||||||||||||||||||||||||||||||||||||||
| InChI Key: | QYHFIVBSNOWOCQ-UHFFFAOYSA-N | ||||||||||||||||||||||||||||||||||||||||||||||||
| InChI: | InChI=1S/H2O4Se/c1-5(2,3)4/h(H2,1,2,3,4) | ||||||||||||||||||||||||||||||||||||||||||||||||
| CAS number: | 7783-08-6 | ||||||||||||||||||||||||||||||||||||||||||||||||
| IUPAC Name: | selenic acid | ||||||||||||||||||||||||||||||||||||||||||||||||
| Traditional IUPAC Name: | selenic acid | ||||||||||||||||||||||||||||||||||||||||||||||||
| SMILES: | O[Se](O)(=O)=O | ||||||||||||||||||||||||||||||||||||||||||||||||
| Chemical Taxonomy | |||||||||||||||||||||||||||||||||||||||||||||||||
| Taxonomy Description | This compound belongs to the class of inorganic compounds known as non-metal selenates. These are inorganic non-metallic compoundscontaining a selenate as its largest oxoanion. | ||||||||||||||||||||||||||||||||||||||||||||||||
| Kingdom | Inorganic compounds | ||||||||||||||||||||||||||||||||||||||||||||||||
| Super Class | Mixed metal/non-metal compounds | ||||||||||||||||||||||||||||||||||||||||||||||||
| Class | Other mixed metal/non-metal oxoanionic compounds | ||||||||||||||||||||||||||||||||||||||||||||||||
| Sub Class | Non-metal selenates | ||||||||||||||||||||||||||||||||||||||||||||||||
| Direct Parent | Non-metal selenates | ||||||||||||||||||||||||||||||||||||||||||||||||
| Alternative Parents | |||||||||||||||||||||||||||||||||||||||||||||||||
| Substituents |
| ||||||||||||||||||||||||||||||||||||||||||||||||
| Molecular Framework | Acyclic compounds | ||||||||||||||||||||||||||||||||||||||||||||||||
| External Descriptors |
| ||||||||||||||||||||||||||||||||||||||||||||||||
| Physical Properties | |||||||||||||||||||||||||||||||||||||||||||||||||
| State: | Not Available | ||||||||||||||||||||||||||||||||||||||||||||||||
| Charge: | -2 | ||||||||||||||||||||||||||||||||||||||||||||||||
| Melting point: | Not Available | ||||||||||||||||||||||||||||||||||||||||||||||||
| Experimental Properties: |
| ||||||||||||||||||||||||||||||||||||||||||||||||
| Predicted Properties |
| ||||||||||||||||||||||||||||||||||||||||||||||||
| Biological Properties | |||||||||||||||||||||||||||||||||||||||||||||||||
| Cellular Locations: | Cytoplasm | ||||||||||||||||||||||||||||||||||||||||||||||||
| Reactions: | |||||||||||||||||||||||||||||||||||||||||||||||||
| Pathways: |
| ||||||||||||||||||||||||||||||||||||||||||||||||
| Spectra | |||||||||||||||||||||||||||||||||||||||||||||||||
| Spectra: |
| ||||||||||||||||||||||||||||||||||||||||||||||||
| References | |||||||||||||||||||||||||||||||||||||||||||||||||
| References: | Not Available | ||||||||||||||||||||||||||||||||||||||||||||||||
| Synthesis Reference: | Not Available | ||||||||||||||||||||||||||||||||||||||||||||||||
| Material Safety Data Sheet (MSDS) | Not Available | ||||||||||||||||||||||||||||||||||||||||||||||||
| Links | |||||||||||||||||||||||||||||||||||||||||||||||||
| External Links: |
| ||||||||||||||||||||||||||||||||||||||||||||||||
Enzymes
- General function:
- Involved in oxidoreductase activity
- Specific function:
- The nitrate reductase enzyme complex allows Pseudomonas aeruginosa to use nitrate as an electron acceptor during anaerobic growth. The alpha chain is the actual site of nitrate reduction
- Gene Name:
- narG
- Locus Tag:
- PA3875
- Molecular weight:
- 141 kDa
Reactions
| Nitrite + acceptor = nitrate + reduced acceptor. |
- General function:
- Involved in iron-sulfur cluster binding
- Specific function:
- The nitrate reductase enzyme complex allows Pseudomonas aeruginosa to use nitrate as an electron acceptor during anaerobic growth. The beta chain is an electron transfer unit containing four cysteine clusters involved in the formation of iron-sulfur centers. Electrons are transferred from the gamma chain to the molybdenum cofactor of the alpha subunit
- Gene Name:
- narH
- Locus Tag:
- PA3874
- Molecular weight:
- 58.1 kDa
Reactions
| Nitrite + acceptor = nitrate + reduced acceptor. |
- General function:
- Involved in nitrate reductase activity
- Specific function:
- The nitrate reductase enzyme complex allows Pseudomonas aeruginosa to use nitrate as an electron acceptor during anaerobic growth. The gamma chain is a membrane-embedded heme-iron unit resembling cytochrome b, which transfers electrons from quinones to the beta subunit
- Gene Name:
- narI
- Locus Tag:
- PA3872
- Molecular weight:
- 25 kDa
Reactions
| Nitrite + acceptor = nitrate + reduced acceptor. |
- General function:
- Involved in catalytic activity
- Specific function:
- ATP + sulfate = diphosphate + adenylyl sulfate
- Gene Name:
- cysD
- Locus Tag:
- PA4443
- Molecular weight:
- 35.5 kDa
Reactions
| ATP + sulfate = diphosphate + adenylyl sulfate. |
- General function:
- Involved in GTPase activity
- Specific function:
- May be the GTPase, regulating ATP sulfurylase activity
- Gene Name:
- cysN
- Locus Tag:
- PA4442
- Molecular weight:
- 69.3 kDa
Reactions
| ATP + sulfate = diphosphate + adenylyl sulfate. |
- General function:
- Involved in oxidoreductase activity
- Specific function:
- Catalytic subunit of the periplasmic nitrate reductase (NAP). Only expressed at high levels during aerobic growth. NapAB complex receives electrons from the membrane-anchored tetraheme protein napC, thus allowing electron flow between membrane and periplasm. Essential function for nitrate assimilation and may have a role in anaerobic metabolism
- Gene Name:
- napA
- Locus Tag:
- PA1174
- Molecular weight:
- 92.9 kDa
Reactions
| Nitrite + acceptor = nitrate + reduced acceptor. |
- General function:
- Energy production and conversion
- Specific function:
- Small subunit of the periplasmic nitrate reductase (NAP). Only expressed at high levels during aerobic growth. NapAB complex receives electrons from the membrane-anchored tetraheme napC protein, thus allowing electron flow between membrane and periplasm. Essential function for nitrate assimilation and may have a role in anaerobic metabolism
- Gene Name:
- napB
- Locus Tag:
- PA1173
- Molecular weight:
- 17.9 kDa
- General function:
- Involved in unfolded protein binding
- Specific function:
- Chaperone required for proper molybdenum cofactor insertion and final assembly of the membrane-bound respiratory nitrate reductase 1. Required for the insertion of the molybdenum into the apo-NarG subunit, maybe by keeping NarG in an appropriate competent-open conformation for the molybdenum cofactor insertion to occur. NarJ maintains the apoNarGH complex in a soluble state. Upon insertion of the molybdenum cofactor, NarJ seems to dissociate from the activated soluble NarGH complex, before its association with the NarI subunit on the membrane
- Gene Name:
- narJ
- Locus Tag:
- PA3873
- Molecular weight:
- 27.3 kDa