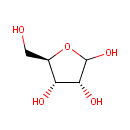
Ribose (PAMDB000116)
| Record Information | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Version | 1.0 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Update Date | 1/22/2018 11:54:54 AM | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Metabolite ID | PAMDB000116 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Identification | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Name: | Ribose | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Description: | Ribose is a pentose that is actively used in many biological systems usually in its D-form. Ribose constitutes the backbone of RNA, a biopolymer that is the basis of genetic transcription. It is related to deoxyribose, as found in DNA. Once phosphorylated, ribose can become a subunit of ATP, NADH, and several other compounds that are critical to metabolism like the secondary messengers cAMP and cGMP. In cells D-ribose must be phosphorylated before it can be used. Ribokinase catalyzes this reaction by converting D-ribose to D-ribose 5-phosphate. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Structure | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Synonyms: |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Chemical Formula: | C5H10O5 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Average Molecular Weight: | 150.1299 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Monoisotopic Molecular Weight: | 150.05282343 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| InChI Key: | HMFHBZSHGGEWLO-SOOFDHNKSA-N | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| InChI: | InChI=1S/C5H10O5/c6-1-2-3(7)4(8)5(9)10-2/h2-9H,1H2/t2-,3-,4-,5?/m1/s1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| CAS number: | 50-69-1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| IUPAC Name: | (3R,4S,5R)-5-(hydroxymethyl)oxolane-2,3,4-triol | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Traditional IUPAC Name: | D-ribofuranoside | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| SMILES: | OC[C@H]1OC(O)[C@H](O)[C@@H]1O | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Chemical Taxonomy | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Taxonomy Description | This compound belongs to the class of organic compounds known as monosaccharides. These are compounds containing one carbohydrate unit not glycosidically linked to another such unit, and no set of two or more glycosidically linked carbohydrate units. Monosaccharides have the general formula CnH2nOn. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Kingdom | Organic compounds | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Super Class | Organooxygen compounds | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Class | Carbohydrates and carbohydrate conjugates | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Sub Class | Monosaccharides | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Direct Parent | Monosaccharides | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Alternative Parents | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Substituents |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Molecular Framework | Aliphatic heteromonocyclic compounds | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| External Descriptors |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Physical Properties | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| State: | Not Available | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Charge: | 0 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Melting point: | Not Available | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Experimental Properties: |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Predicted Properties |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Biological Properties | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Cellular Locations: | Cytoplasm | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Reactions: | Adenosine triphosphate + Water + Ribose > ADP + Hydrogen ion + Phosphate + Ribose Adenosine triphosphate + Water + Ribose > ADP + Hydrogen ion + Phosphate + Ribose Cytidine + Water > Cytosine + Ribose Water + Uridine > Ribose + Uracil Adenosine + Water > Adenine + Ribose Water + Inosine > Hypoxanthine + Ribose Water + Xanthosine > Ribose + Xanthine Water + D-Ribose-5-phosphate > Phosphate + Ribose Adenosine triphosphate + Ribose <> ADP + Hydrogen ion + D-Ribose-5-phosphate Adenosine triphosphate + Ribose <> ADP + D-Ribose-5-phosphate Guanosine + Water <> Guanine + Ribose A pyrimidine nucleoside + Water > Ribose + a pyrimidine base Pyrimidine nucleoside + Water <> Ribose + Pyrimidine | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Pathways: | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Spectra | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Spectra: | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| References | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| References: |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Synthesis Reference: | Park, Yong-Cheol; Choi, Jin-Ho; Bennett, George N.; Seo, Jin-Ho. Characterization of D-ribose biosynthesis in Bacillus subtilis JY200 deficient in transketolase gene. Journal of Biotechnology (2006), 121(4), 508-516. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Material Safety Data Sheet (MSDS) | Download (PDF) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Links | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| External Links: |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Enzymes
- General function:
- Involved in nucleotide binding
- Specific function:
- Part of the ABC transporter complex RbsABCD involved in ribose import. Responsible for energy coupling to the transport system
- Gene Name:
- rbsA
- Locus Tag:
- PA1947
- Molecular weight:
- 55.8 kDa
Reactions
| ATP + H(2)O + monosaccharide(Out) = ADP + phosphate + monosaccharide(In). |
- General function:
- Involved in phosphotransferase activity, alcohol group as acceptor
- Specific function:
- ATP + D-ribose = ADP + D-ribose 5-phosphate
- Gene Name:
- rbsK
- Locus Tag:
- PA1950
- Molecular weight:
- 31.8 kDa
Reactions
| ATP + D-ribose = ADP + D-ribose 5-phosphate. |
- General function:
- Involved in acid phosphatase activity
- Specific function:
- Dephosphorylates several organic phosphomonoesters and catalyzes the transfer of low-energy phosphate groups from phosphomonoesters to hydroxyl groups of various organic compounds. Preferentially acts on aryl phosphoesters. Might function as a broad-spectrum dephosphorylating enzyme able to scavenge both 3'- and 5'-nucleotides and also additional organic phosphomonoesters
- Gene Name:
- aphA
- Locus Tag:
- PA1409
- Molecular weight:
- 38 kDa
Reactions
| A phosphate monoester + H(2)O = an alcohol + phosphate. |
- General function:
- Involved in transporter activity
- Specific function:
- Part of the binding-protein-dependent transport system for ribose. Probably responsible for the translocation of the substrate across the membrane
- Gene Name:
- rbsC
- Locus Tag:
- PA1948
- Molecular weight:
- 34 kDa
- General function:
- Carbohydrate transport and metabolism
- Specific function:
- Involved in the high-affinity D-ribose membrane transport system and also serves as the primary chemoreceptor for chemotaxis
- Gene Name:
- rbsB
- Locus Tag:
- PA1946
- Molecular weight:
- 33.9 kDa
Transporters
- General function:
- Involved in nucleotide binding
- Specific function:
- Part of the ABC transporter complex RbsABCD involved in ribose import. Responsible for energy coupling to the transport system
- Gene Name:
- rbsA
- Locus Tag:
- PA1947
- Molecular weight:
- 55.8 kDa
Reactions
| ATP + H(2)O + monosaccharide(Out) = ADP + phosphate + monosaccharide(In). |
- General function:
- Involved in transporter activity
- Specific function:
- Part of the binding-protein-dependent transport system for ribose. Probably responsible for the translocation of the substrate across the membrane
- Gene Name:
- rbsC
- Locus Tag:
- PA1948
- Molecular weight:
- 34 kDa
- General function:
- Carbohydrate transport and metabolism
- Specific function:
- Involved in the high-affinity D-ribose membrane transport system and also serves as the primary chemoreceptor for chemotaxis
- Gene Name:
- rbsB
- Locus Tag:
- PA1946
- Molecular weight:
- 33.9 kDa